QbSyst2e.14.html
Système du Québécium
Suivant QbSyst2e.15.html
Précédent
QbSyst2e.13.htmlDébut QbSyst2e.1.html
Analyse des tableaux d'anticodons dans la pseudo strate 3.
Remarques générales.
Dans les figures 156 à 171, la pseudo strate démontre son utilité pour présenter de façon ordonnée les anticodons de tous les tableaux accessibles, qui appartiennent à 5 codes biomoléculaires : cb nomal, cb2, cb4, cb5 et cb12. Les 48 cases de la grille suffisent pour les loger, y compris les doubles emplois. Voyez les figures 124 et 155, les classes O et E d'anticodons.Classe O. Le plus souvent, remplissage exact des 16 cases du bloc central, parfois, il reste des cases vides. Dans l'un des tableaux, 3 doubles emplois de cette classe occupent en outre 3 cases supplémentaires (H. sapiens).
Classe E. Le nombre de cases employées est fortement variable et va de 6 à 32
Des symétries EO plus ou moins parfaites se manifestent dans la pseudo strate 3, quant à l'occupation des cases et aux multiplicités qui y apparaissent. Voyez par exemple E. coli (Lecomte) et U. urealyticum (figures 156 et 163). Les mitochondries des mammifères et de Limulus se signalent par leur économie d'anticodons : 22 seulement, dont 6 hors bloc central; en retour, chacun de ces anticodons reconnaît en moyenne un nombre relativment élevé de codons.
Statistiques. Voici un relevé des nombres d'anticodons dans la pseudo strate 3, en référence aux figures 156 à 171. Tableau 43. Les nombres d'anticodons inscrits vont de 22 à 48, moyenne 34,4, si l'on excepte les doubles emplois et de 22 à 48, moyenne 35,8, si l'on inclut les doubles emplois.
.......................................
Tableau 43...........................
Nombre d'anticodons.....................
Anticodons dans la pseudo strate 3Source
.....Antic. tot =.Classe O.+Classe E.....D. empl......cbLim.mito St
....22................16................6..............0......cb5Mmit Ssbg**
...22...............16.................6..............0......cb2Mc uab
............28...............10...............18..............1......cb4A.t.plast. T
....29................16...............13..............1......cbnormalU.u. uab
..........30...............10...............20..............0......cb4A.t.mito. T
..... 31...............16...............15..............4......cbnormalMaloy
............32...............16...............16..............0......cbnormalMp Mg uab*.
...32...............12...............20..............1......cb4E. coli Lec.
......36..............16................20.............0.....cbnormalA.t.cyto. T
......37...............16...............21.............8......cbnormalS.cer. Mun
.......38...............16...............22.............2.....cbnormalC. albic.
..........39...............15...............24.............0.....cb12E.coli uab
........40...............16...............24.............1......cbnormalEuc. Lec
...........45...............16...............29.............0.....cbnormalH.sap. Nat
.....45...............16...............29.............3.....cbnormalC. ele. L
..........45...............16...............29.............1.....cbnormalSomme et moyenne doubles emplois
............. ....22/16 = 1,4Sommes et moyennes sans les doubles emplois
................................
239/16 = 14,9..............
551/16 = 34,4.....................312/16 = 19,5Ycompris les doubles emploi
.............
573/16 = 35,8*2 espèces
** 31 espèces. Tableau 44. 31 espèces de mammifères, dont l'être humain, le rhinocéros et le rorqual bleu Balaenoptera musculus, le plus gros animal connu. http://lesbaleines.net/rorqualbleu.htm
Tableau 44
31 espèces de mammifères
http://mamit-trna.u-strasbg.fr/Sequences.html
Homo sapiens
...............Gorilla gorillaPan paniscus
...............Pan troglodytesPongo pygmaeus
...............Hylobates larPapio hamadryas
...............Equus caballusEquus asinus
...............Rhinoceros unicornisCeratotherium simum
...............Bos taurusOvis aries
...............Sus scrofaBalaenoptera musculus
...............Balaenoptera physalusHippopotamus amphibius
...............Mus musculusRattus norvegicus
...............Myoxus glisOryctolagus cuniculus
...............Felis catusHalichoerus grypus
...............Phoca vitulinaCanis familiaris
...............Artibeus jamaicensisErinaceus europeus
...............Dasypus novemcinctusDidelphis virginiana
...............Macropus robustusOrnithorhyncus anatinus
Escherichia coli. pStrate 3 modèle?
Les anticodons d'Escherichia coli sont au nombre de 36 dans le tableau de Lecomte. Ils entrent exactement dans une strate 3 de 36 cases, et c'est le seul cas d'une strate 3 de 36 cases. (Figures 139, 156). Le total de 36 se répète pour Neisseria meningitis MC58 (TIGR), Chlamydia pneumoniae J138 (Japon), Chlamydia pneumoniae AR39 (TIGR), Chlamydia pneumoniae CML029 (UCB) dans le tableau des anticodons ci-dessous. Tableau 45.
D'après les données disponibles, la strate 3 avec ses 36 cases ordonnées ne constitue évidemment pas une exigence pour les anticodons. Elle paraît cependant représenter une sorte de modèle réalisé approximativement et en moyenne. Voici la liste ordonnée des 16 nombres d'anticodons O et E du Tableau 43. Moyenne 34,4, médiane 34.
22, 22, 28, 29; 30, 31, 32, 32; 36, 37, 38, 39; 40, 45, 45, 45
La diversité de ces nombres semble être une règle. Leur distribution n'évoque nullement une courbe de Gauss mais suggère plutôt une bimodalité avec rassemblement aux valeurs extrêmes 45 et 22. La valeur 22 pourrait apparaître 32 fois au lieu de 2, si l'on comptait chacune des espèces du Tableau 44, formant une distribution aiguille à 22 anticodons.
Analyse génomique.
Nous utilisons les listes d'anticodons de Todd Lowe. Ses listes ne donnent cependant pas les codons reconnus par les anticodons. Par suite, ses résultats ne se prêtent pas commodément à l'inscription dans la grille de la strate 4 et de la pseudo strate 3. Il se prêtent toutefois au dénombrement des anticodons, ce qui est fait ci-dessous. Tableau 45.
Tableau 45
Nombre d"anticodons.
Tableau des anticodons génomiques
http://rna.wustl.edu/GtRDB/
D'après TD. Mise en ordre numérique.
Acides aminés normaux (+ présence de Sélénocystéine)
Bactéries
..............................Chlamydia trachomatis MoPn (TIGR)
............... 26Ureaplasma urealyticum
............... 27+Staphylococcus aureus N315
............... 28Borrelia burgdorferi
............... 29Buchnera sp.
............... 29Rickettsia prowazekii
............... 30Bacillus halodurans
............... 31Pasteurella multocida
............... 31+Haemophilus influenzae Rd
............... 32+Campylobacter jejuni
............... 32+Vibrio cholerae
............... 32Mycoplasma genitalium
............... 33+Mycoplasma pneumoniae.....
............... 33+Helicobacter pylori 26695 (TIGR)
............... 33Helicobacter pylori J99 (Astra)
............... 33Bacillus subtilis
............... 33Chlamydia trachomatis D/UW-3/Cx (UCB)
............... 34Neisseria meningitidis Z2491 (Sanger)
............... 34Chlamydia pneumoniae CWL029 (UCB)
............... 36Chlamydia pneumoniae AR39 (TIGR)
............... 36Chlamydia pneumoniae J138 (Japan)
............... 36Neisseria meningitidis MC58 (TIGR)
............... 36Pseudomonas aeruginosa
............... 38+Synechocystis sp.
............... 39Escherichia coli K-12
............... 39+Aquifex aeolicus
............... 39+Escherichia coli O157:H7 EDL933 (Wisconsin)
............... 40+Escherichia coli O157:H7 RIMD 0509952 (Osaka)
............... 40+Caulobacter crescentus
............... 41Deinococcus radiodurans
............... 42Mycobacterium tuberculosis
............... 43Treponema pallidum
............... 43Xylella fastidiosa
............... 43Mycobacterium leprae
............... 43Thermotoga maritima
............... 44Somme et meyenne 1238/35 = 35,4
Archaea
..............................Methanococcus maripaludis
............... 31+Methanococcus jannaschii
............... 32+Methanobacterium thermoautotrophicum
............... 34Thermoplasma acidophilum
............... 43Thermoplasma volcanum
............... 43Aeropyrum pernix
............... 43Sulfolobus solfataricus
............... 43Sulfolobus tokodaii
............... 43Archaeoglobus fulgidus
............... 44Pyrococcus horikoshii
............... 44Pyrococcus furiosus
............... 44Pyrococcus abyssi
............... 44Halobacterium sp.
............... 44Pyrobaculum aerophilum
............... 44Methanosarcina mazei
...............44Somme et moyenne 520/15 = 41,3
Eucaryotes
..............................Saccharomyces cerevisiae
............... 41Drosophila melanogaster
............... 43+Schizosaccharomyces pombe
............... 45Caenorhabditis elegans
............... 46+Homo sapiens
............... 48+Arabidopsis thaliana
............... 48Somme et moyenne 271/6 = 45,2
Somme et moyenne globales 2129/56 = 38,0
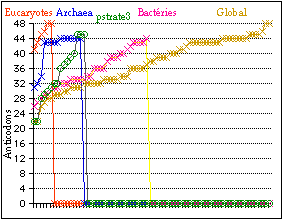
Les résultats des dénombrements sont montrés au Tableau 46 et à la Figure 172. Les Eucaryotes ont le plus grand nombre moyen d'anticodons (45,2), puis viennent dans l'ordre les Archaea (41,3) et les Bactéries (35,4). La pseudo strate 3 présente un nombre inférieur (34,4), qui tient surtout à l'inclusion des anticodons mitochondriaux qui sont au nombre de 22 pour les mammifères et pour un invertébré.
Tableau 46.
Nombre d"anticodons.
Sommaire des Tableaux 43 et 45.
Anticodons dans la pseudo strate 3
...............Somme et moyenne 551/16 = 34,4
Bactéries TD
...................................Somme et moyenne 1238/35 = 35,4
Archaea TD
...................................Somme et moyenne 520/15 = 41,3
Eucaryotes TD
...................................Somme et moyenne 271/6 = 45,2
Les 4 séries
...................................Somme et moyenne globales 2680/72 = 37,2
Moyenne des valeurs extrêmes (22+48)/2 = 35
Médiane 38

Fig. 172. Vue d'ensemble selon 72 tableaux d'anticodons d'après le Tableau 46.
Conclusion.
À suivre.
Les anticodons sont des triplets qui n'existent pas isolés ni rangés à la queue leu leu comme les triplets des codons dans la chaîne de l'ARNm. Ils n'ont d'existence que enserrés dans une séquence d'ARNt et d'efficacité qu'engagés dans la masse d'un ribosome. La suite de cette étude demanderait d'examiner les séquences et les ribosomes.Il y a 36 et 36. Voilà donc une 2e fois que le nombre 36 se trouve associé aux anticodons. Précédemment, nous avons signalé qu'ils occupent les positions 34, 35 et 36 dans la séquence de l'ARNt. Figures 54, 55, 56. QbSyst2e.06.html
Strate 3 de 36 anticodons. Voici un essai de théorie. Une strate 3 de 36 anticodons serait une sorte de modèle anti ou antimodèle, ne devant être réalisé qu'avec un grand écart, selon 2 règles :
....................
il est obligatoire de la réaliser en moyenne; les écarts vont de -14 pour les mitochondries animales à +12 pour certains Eucaryotes;....................
il est obligatoire de ne pas la réaliser exactement ou avec un faible écart.La figure 156 peut nous servir d'antimodèle, même si les résultats des Tableaux 43 et 45 ne la confirment pas pour Escherichia coli.
Exemple d'un antimodèle : le centre de gravité d'un tore. Il est à la fois significatif et dépourvu de matière.
Contraste numérique. Répétons-le : le contraste est tout à fait remarquable entre les nombres des codons et des acides aminés normaux, valant constamment 64+0 -0 et 20+0 -0 respectivement, et les nombres des anticodons valant 36+12 -14.
Strate 3p de 48 cases. La pseudo strate 3 de 48 cases ou strate 3p résulte d'une adpatation du système du québécium. Quoiqu'il en soit de son interprétation théorique, elle est un formalisme qui jusqu'ici a permis d'afficher d'une façon originale et pratique les anticodons de tous les codes biomoléculaires rencontrés.
Codes biomoléculaires 4 strates.
Le formalisme de la strate 3p permet encore de constituer des codes biomoléculaires complets en 4 strates. Dans le Chapitre 4, nous avons répertorié 16 codes biomoléculaires de 3 strates, tenant compte des dérogations au code normal. QbSyst2e.8.html
Les nouveaux codes en 4 strates augmenteront ce répertoire, parce qu'en général un code 3 strate donné est associé à plusieurs sortes de strates 3p, comme le montre le Tableau 43.
Les figures 173 et 174 donnent des aperçus de tels codes biomoléculaires 4 strates.

Fig. 173 Code biomoléculaire de 4 strates et 132 cases.
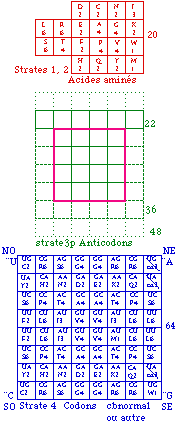
Fig. 174. Aperçu d'un code biomoléculaire 4 strates. De la strate 3p on montre le bloc central et des nombres représentatifs d'anticodons.
Mais avant de confectionner de tels codes, il nous faut passer aux codenzymes.
QbSyst2e.14.html
Système du Québécium
Suivant QbSyst2e.15.html
Précédent
QbSyst2e.13.htmlDébut QbSyst2e.1.html