QbSyst2e.11.html
Système du Québécium
Suivant QbSyst2e.12.html
Précédent
QbSyst2e.10.htmlDébut QbSyst2e.1.html
Chapitre 7
Applications aux biomolécules
Anticodons et codenzymes
Le code enzymatique

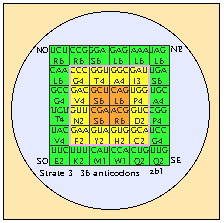
Fig. 111. Les anticodons d'Escherichia coli (d'après Lecomte).
61 anticodons? 20 anticodons?
Dans le cadre de la présente étude, l'hypothèse séduisante se propose d'elle-même, que les anticodons, intermédiaires biologiques caractérisés entre 64 codons et 20 acides aminés, soient aussi intermédiaires numériquement entre 64 et 20. Plus précisément, ils s'ajusteraient à 36, nombre prévu pour la strate 3 intermédiaire entre 64 et 20.
Nous allons chercher dans quelle mesure la réalité se rapproche de cette hypothèse. Figure 112.
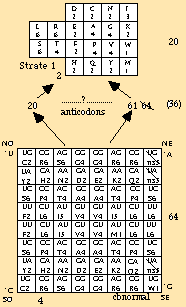
Fig. 112. Le nombre des anticodons : entre 20 et 61 dans le code normal. 36 cases dans la strate 3.
À cette fin, nous allons établir un protocole d'analyse et de présentation des données.
L'expérience a montré qu'il n'y a pas un nombre unique d'anticodons, alors qu'il y a un nombre unique d'acides aminés normaux (20) et un nombre unique de codons (64). Leur nombre varie selon les espèces, à l'intérieur d'une espèce et selon les souches.
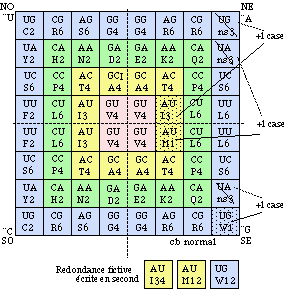
Fig . 113. Redondances fictives écrites en second. 64 cases fictives.
Arithmétique. Redondances fictives. 64 cases codantes fictives.
Parification. Dans les cases de redondance impaire de la strate 4, nous montons la redondance à la valeur paire supérieure et nous imaginons une case fictive supplémentaire. La redondance fictive paire s'écrit après l'impaire. Pour le code normal, cette opération affecte les cases codantes de 3 acides aminés :
I3 devient I34,
M1 devient M12,
W1 devient W12.
Nous créons ainsi 3 cases codantes fictives qui peuvent remplacer les 3 cases stop. Le nombre (fictif) de cases codantes dans le code normal devient 64. Figure 113. Telle parification s'appliquera aussi bien aux autres codes.
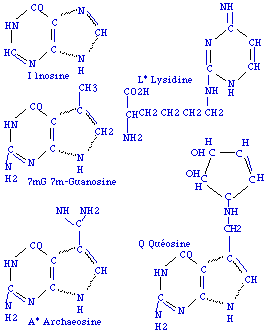
Fig. 114. I, Inosine.- L*, Lysidine, qui est acide aminé par une extrémité . - 7mG, 7-méthyl-Guanosine.- Q, Quéosine.- A*, Archaeosine.
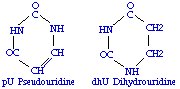
Fig. 115. pU,
yU, pseudo-Uridine. - dhU, Dihydrouridine.
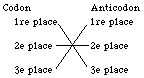
Fig. 116. Écriture. Places des bases correspondantes dans l'écriture du codon et de l'anticodon.
Données expérimentales.
En 1er lieu : les correspondances UA, CG entre les bases des codons et des anticodons servent comme une étape initiale et sont complétées par une maturation qui introduit des bases azotées nouvelles dans les anticodons. Par exemple, la maturation remplacera A par I. Figures 114, 115,Tableau 41.
En 2e lieu, une convention d'écriture : la base en 1re place du codon correspond à celle écrite en 3e place de l'anticodon. La base en 3e place du codon correspond à celle écrite en 1re place de l'anticodon. Il y a une correspondance croisée. Figure 116.
En 3e lieu, il y a le flottement. Entre la base écrite en 3e place du codon et la base qui lui correspond dans l'anticodon, il n'y a pas de correspondance uni-univoque. C'est ce qu'on appelle le flottement. Il y a plusieurs possibilités, partiellement exposées dans la figure 117. La 1re base écrite dans l'anticodon est la base flottante.
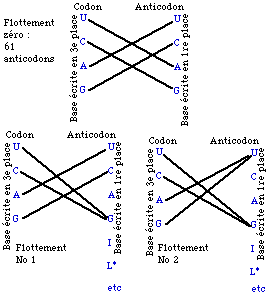
Fig. 117.Flottement. Correspondances entre la base écrite en 3e place dans le codon et la base écrite en 1re place dans l'anticodon. 3 hypothèses de flottement.
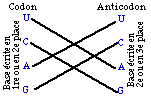
Fig. 118. Pas de flottement. Correspondances exactes pour les bases autres que celles de la figure précédente.
Tableau 41. Nouvelles bases azotées des anticodons.
I, Inosine,
L*, Lysidine,
7mG, 7méthyl-Guanosine,
Q, Quéosine,
A*, Archaeosine,
pU,
yU, pseudo-Uridine,dhU, Dihydrouridine
xo5U, hydroxy-méthyl-Uridine, dérivé,
xm5U, méthyl-Uridine, dérivé,
Il n'y a pas de flottement entre les bases écrites aux autres places. Figure 118.
Un flottement zéro donnerait 61 anticodons.
Essayons d'appliquer la règle de flottement No 2 à 64 codons fictifs : puisque chaque codon fait partie d'une paire soit (UC) soit (AG), le résultat est 32 anticodons. La règle de flottement No 1 donnerait plus que 32 anticodons.
Strate 4 condensée.
Voici une modification de la grille de la strate 4 qui nous servira. D'abord pour le code normal cb normal.
Pairages NS. D'après les symétries discutées au chapitre 3, les cases NO et SO forment invariablement des paires (UC) codant pour le même acide aminé.
Des paires comparables (AG) se rencontrent, mais moins souvent, entre les cases NE et SE.Primo. Nous rassemblons le contenu de chacune des paires de cases (UC) dans une seule du quadrant NO. Cela signifie que nous replions le quadrant SO sur le quadrant NO. Ainsi, UGC sera écrit dans la même case que UGU dans le quadrant NO. Le quadrant SO disparaît. Figure 119.
C'est une opération de symétrie miroir par rapport à l'axe horizontal de la strate 4. 32 codons pairés (UC) sont écrits dans 16 cases. Nous avons 48 cases en 8 colonnes. L'ordonnance des 1re et 2e lettres est préservée.
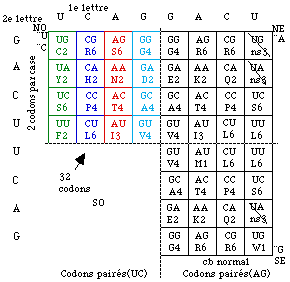
Fig. 119. Condensation de la strate 4. Primo. Le quadrant SO est disparu.
Secundo. Nous alignons les 16 cases O en 2 colonnes. Nous obtenons 48 cases en 6 colonnes après des échanges portant sur 8 cases. Figure 122.
Partout est préservée l'ordonnance de la 2e lettre. Quant à la 1re lettre, son ordonnance est préservée dans la moitié E; partout est préservée l'ordonnance de son noyau, pyrimidique UC ou purique AG. Et voilà la strate 4 condensée en 48 cases et 6 colonnes. Elle a un demiant O et 2 quadrants NE, SE. Figures 120,121.
La strate 4 condensée nous rendra service. Elle ressemble à la strate 3 par le nombre de colonnes (6) mais a 2 rangéees de plus. Elle a le même contenu de 64 codons que la strate 4 habituelle. Figures 121, 122.
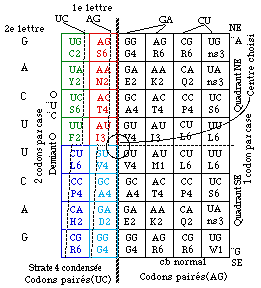
Fig. 120. Secundo. Comme la figure précédente, mais placements remaniés dans le demiant O, ajoutant aux symétries. De la sorte, la 1re lettre du codon dans une colonne conserve le noyau pyrimidine UC ou le noyau purine AG.
Nous appelons demiant un espace de 180o autour du centre choisi. La création du demiant O répond à la présomption que les codons de chacune de ses cases n'interviendront que pairés, les 2 codons d'une paire étant reconnus par un même anticodon.
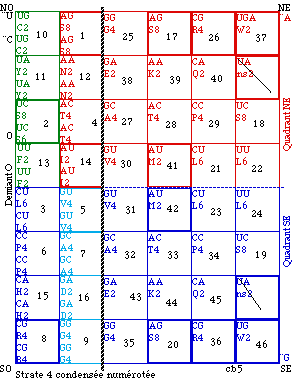
Ordre de lecture. Nous établissons un ordre de lecture des cases dans la strate 4 condensée. D'abord le demiant O, puis les quadrants E. Dans l'ordre de lecture habituel, de gauche à droite et de haut en bas. Nous numérotons les cases d'après leurs redondances fictives. les plus élevées d'abord, soit dans l'ordre toutes les cases 6, puis 4, puis 2. C'est la strate 4 condensée numérotée. Figure 122. Puis nous réarrangeons les cases dans l'ordre de la lecture. C'est la strate 4 condensée ordonnée. Figure 123.
Voici dans quel ordre les divers acides aminés apparaissent pour la 1e fois dans la lecture. Pour 15 d'entre eux, cela se produit dans la lecture du demiant O. Tableau 42.
Les 16 cases du demiant O présentent peu de répétitions : 15 apparitions d'acides aminés pour 16 cases. Seule répétition : S6 qui occupe les cases 1 et 2. En revanche, les quadrants E apportent plusieurs répétitions (24) et seulement 5 apparitions.
Dissymétrie Est Ouest.
La réalisation de la modificaton décrite de la strate 4 donne l'occasion de remarquer la dissymétrie qui existe entre demiants Est et Ouest de cette strate du code normal. L'opération primo effectuée sur le demiant Est n'aurait pas le même intérêt, puisque le pairage entre les cases des quadrants Nord et Sud est imparfait à cause des 3 cases stop, des cases isolées I, M et W.
Cette dissymétrie résulte évidemment des différences des rôles entre bases écrites en 3e place des codons : puriques dans le quadrant Est, pyrimidiques dans le quadrant Ouest.
Code 2 et 5 en 32 cases. Un code biomoléculaire primitif restreint.
Il en est autrement pour les codes 2 et 5 mitochondriaux des vertébrés et des invertébrés respectivement, où le pairage Nord Sud est exact partout (si l'on néglige la différence entre M et fM). Par repli autour d'un axe horizontal OE, on peut alors dessiner une strate 4 condensée en 32 cases dont chacune renferme 2 codons de même rôle. (Figures 127, 133). On peut voir là une étape pour imaginer un code biomoléculaire primitif ayant moins que 64 codons.
Tableau 42. Apparition des 20 acides aminés dans la lecture de la strate 4 condensée. cb normal.
Demiant O, 16 cases codantes
S6 1
L6 3
R6 4
T4 5
I34 6
V4 7
P4 8
A4 9
G4 10
C2 11
Y2 12
N2 13
F2 14
H2 15
D2 16
Quadrants E, 29 cases codantes
E2 38
K2 39
Q2 40
M12 41
W12 45
Souligné : redondance vraie, redondance fictive
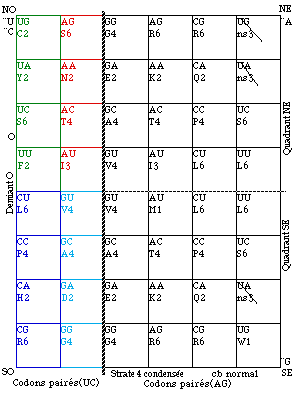
Fig. 121. Strate 4 condensée bis. Chaque case O contient 2 codons pour une entrée. cb normal.
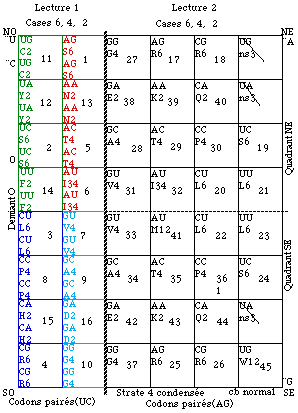
Fig. 122. Strate 4 condensée ter. Ici chaque codon a son entrée distincte dans le demiant O. Ordre de lecture des cases . On choisit les redondances fictives 2, 4.
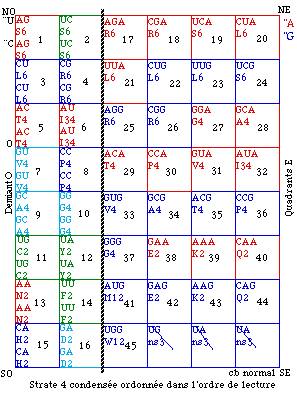
Fig. 123. Strate 4 condensée ordonnée dans l'ordre de lecture. Code normal.
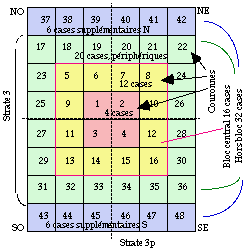
Fig. 124. Pseudostrate 3 de 48 cases sur 6 colonnes, pstrate3, strate 3p. Contient une strate 3 de 36 cases en 3 couronnes et 12 cases supplémentaires. On met en évidence le bloc central de 16 cases. Ordre d'écriture dans les cases 1 à 48. Elle est destinée à recevoir l'écriture des anticodons, un dans chaque case.
Strate 3 allongée. Pseudostrate 3, strate 3p.
À la strate 3 aussi nous allons apporter une modification, cette fois en augmentant le nombre de cases, celles-ci passant de 36 à 48. Nous l'appelons pseudo parce qu'elle ne contient pas exclusivement 36 cases. La pseudo contient une vraie, telle que prévue dans notre système, augmentée symétriquement de cases supplémentaires formant une rangée supérieure et une rangée inférieure. Pour la commodité du langage, nous l'appellerons encore strate 3p, p pour pseudo. La pseudostrate 3 ainsi dessinée est une grille de 48 cases en 6 colonnes aussi bien que la strate 4 condensée mais divisée différemment. Dans une case de la pseudo strate 3, on écrit la formule d'un anticodon, celles des codons qu'il reconnaît et de l'acide aminé qu'il détermine. Figure 124, 125.

Fig. 125. Légende, contenu d'une case de la strate 3p.
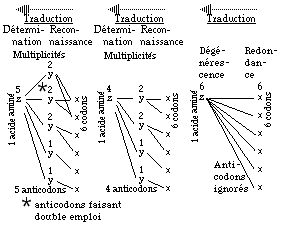
Fig. 126. Redondance, dégénérescences, multiplicités entre codons, anticodons et acides aminés. Double emploi.
Ordre d'écriture dans la strate 3p. Dans le but de pratiquer l'analyse numérique décrite ci-après, on remplit les cases de la pseudo strate dans l'ordre de la figure 124, on suit l'ordre de lecture des cases de la strate 4 condensée ordonnée figures 123. Si un anticodon apparaît plus d'une fois dans la lecture, on ne le lit que la 1re fois. S'il y a un double emploi entre 2 anticodons, on écrit l'un des 2 après tous les autres.
Le procédé consiste essentiellement dans un réarrangement des cases choisies dans la strate 4, placées dans la pseudo strate 3.
pStrates 3. Les caractères numérisables.
Multiplicités. Nous allons reconnaître et analyser les caractères numérisables relatifs à la traduction des codons en acides aminés en passant par les anticodons. Nous nous intéressons aux multiplicités rencontrées et celles-ci se définissent dans un schéma d'arborescence et de graphe. Figure 126.
Dans le code normal, si on ignore l'étape anticodon, la traduction est convergente, plusieurs codons pouvant coder pour un acide aminé. Nous retenons les expressions de redondance des codons et de dégénérence des acides aminés.
Les divergences sont rares, qui correspondraient à une redondance des acides aminés et une dégénérescence des codons. Une divergence notoire est celle du codon AUG, reconnu par l'anticodon CAU, donnant M ou fM.
Si on fragmente la traduction en reconnaissance et détermination, il y a à nouveau des redondances et des dégénérescences que nous appellerons simplement multiplicités, convergentes entre anticodons et acide aminés, mais parfois divergentes entre codons et anticodons.
Dans l'exemple choisi,
................
les 6 codons x ont chacun la redondance 6. L'acide aminé z a la dégénérescence 6.................
L'acide aminé z a la multiplicité 4 ou bien 6;................
les 4 anticodons y ont respectivement les multiplicités 2, 2, 1 et 1, dont la somme est 6;................
ou bien les 5 anticodons y ont respectivement les multiplicités 2, 2, 2, 1 et 1, dont la somme est 8; 2 anticodons font double emploi.Analyse numérique. Les anticodons placés dans les cases de la grille pStrate 3 possèdent plusieurs caractères numérisables pouvant y être inscrits. La figure 126 donne un exemple.
Pour chaque anticodon.
................
Sa position dans la grille, selon les nombres de la figure 126.................
Sa multiplicité soit le nombre de codons qu'il reconnaît.................
Sa multiplicité fictive, égale au nombre fictif, s'il en est, de codons qu'il reconnaît.................
La dégénérescence de l'acide aminé qu'il détermine.................
La dégénérescence fictive, s'il en est, de l'acide aminé qu'il détermine.................
La multiplicité de l'acide aminé qu'il détermine.Pour des ensembles de cases occupées par des anticodons.
Des symétries OE ou autres, ces ensembles pouvant être les couronnes de 4, 12 ou 20 cases, le bloc central de 16 cases ou les 12 cases supplémentaires. Les symétries peuvent toucher une ou plusieurs des multiplicités et des dégénérescences ci-dessus.Codes biomoléculaires 2, 4, 5 et 12.
Certains tableaux d'anticodons qui suivent concernent des codes autres que le code normal décrit plus haut. Figures 119 à 123.
On donne les strates 4 condensées et ordonnées pour les 4 codes qui suivent. Figures 127 à 138.
b2, mitochondrial des vertébrés
cb4, moisissures
cb5 mitochondrial des invertébrés
cb12, levures, autre
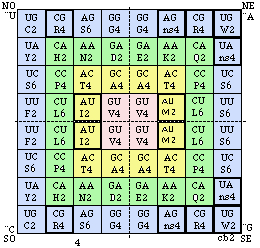
Fig 127 (Fig. 72). cb2 mitochondrial des vertébrés. Comme le code cb normal sauf 12 cases mises en évidence. Strate 4.
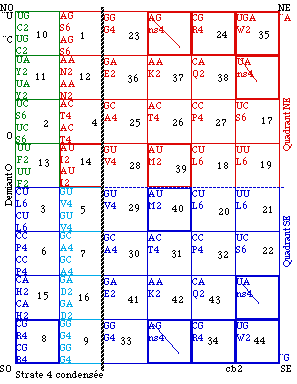
Fig. 128. Strate 4 condensée numérotée dans l'ordre de lecture. Codons et leurs traductions. Comme le code cb normal sauf 11 cases mises en évidence. cb2.
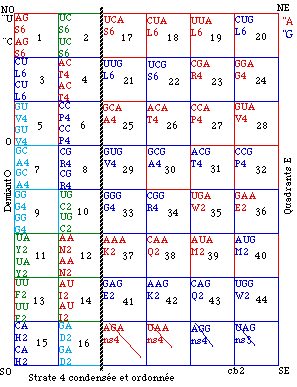
Fig. 129. Strate 4 condensée et ordonnée. cb2.
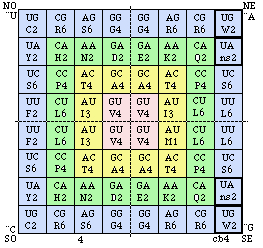
Fig. 130 (Fig. 75). Code biomoléculaire cb4 . Moisissures. Comme le code universel sauf 4 cases mises en évidence. Strate 4
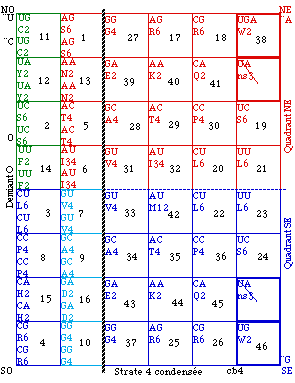
Fig. 131. Strate 4 condensée et numérotée. cb4.
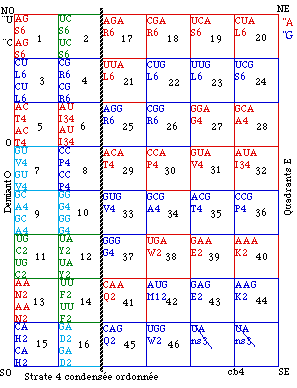
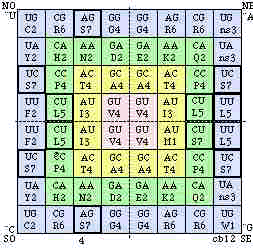
Fig. 132. Strate 4 condensée ordonnée dans l'ordre de lecture. Code cb4. 62 codons codants, 46 cases codantes, 64 codons fictifs.
Fig. 133
(Fig. 76). Code
biomoléculaire cb5. Strate
4. Comme le code normal sauf 18
cases mises en évidence.
Fig. 134 .
Code biomoléculaire cb5.
Strate 4 condensée
numérotée. Comme le
code normal sauf 14 cases mises en
évidence.
Fig. 135.
Code biomoléculaire cb5.
Strate 4 condensée
ordonnée.
Fig. 136
(Fig. 80). cb12. Strate 4. Comme le
code normal sauf 12 cases mises en
évidence.
Fig. 137. cb12.
Strate 4 condensée et
numérotée. Comme le
code normal sauf 9 cases mises en
évidence.
Fig. 138. Strate 4
condensée ordonnée
dans l'ordre de lecture. cb12 Tableaux des
anticodons. Répertoire des
anticodons dans la strate 4.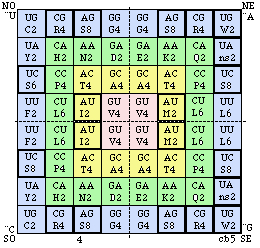



Voici, d'après les données de divers auteurs, les anticodons présentés dans la strate 4 condensée . Figures 139 à 154.
Plus loin, ils sont présentés dans la pseudo strate 3.
Les données des auteurs posent des problèmes d'interprétation, des problèmes relatifs aux parenthèses, aux maturations, aux souches, aux séquences des ARNt porteurs des anticodons et aux doubles emplois. Ces problèmes ne sont pas sans affecter nos résultats. En comparaison, les données sur les codons paraissent mieux établies.
On a admis qu'un même anticodon reconnaît le codon XXU et le codon XXC.
QbSyst2e.11.html
Système du Québécium
Suivant QbSyst2e.12.html
Précédent
QbSyst2e.10.htmlDébut QbSyst2e.1.html